বংশাণু অভিব্যক্তি
এই নিবন্ধে বাংলা পরিভাষাগুলিকে অগ্রাধিকার দেওয়া প্রয়োজন।টির রচনা সংশোধনের প্রয়োজন হতে পারে। কারণ ব্যাকরণ, রচনাশৈলী, বানান বা বর্ণনাভঙ্গিগত সমস্যা রয়েছে। |
বংশাণু অভিব্যক্তি হল এমন প্রক্রিয়া যার মাধ্যমে কোনও বংশাণু (জিন) থেকে প্রাপ্ত তথ্য কার্যকরী বংশাণু উৎপাদ (জিন প্রোডাক্ট) সংশ্লেষণে ব্যবহৃত হয়। এটি আণবিক জীববিদ্যার কেন্দ্রীয় নীতির একটি প্রধান অংশ। এই বংশাণু উৎপাদগুলি প্রায়শই প্রোটিন হলেও, নন-প্রোটিন-কোডিং বংশাণু যেমন ট্রান্সফার আরএনএ (টিআরএনএ) বা স্মল নিউক্লিয়ার আরএনএ (এসএনআরএনএ) বংশাণুর ক্ষেত্রে, ফলাফল হিসেবে কার্যকরী আরএনএ পাওয়া যায় । বংশাণুর অভিব্যক্তি সারসংক্ষেপ প্রকাশিত হয় ১৯৫৮ সালে। ফ্রান্সিস ক্রিক প্রথম কেন্দ্রীয় মতবাদে বংশাণু অভিব্যক্তি নিয়ে আলোচনা করেন। [১] পরবর্তীতে ১৯৭০ সালের প্রবন্ধে এই ধারণা আরো বিকশিত হয়,[২] এবং পরবর্তীতে বিপরীত প্রতিলিপি [৩][৪][৫] এবং আরএনএ প্রতিলিপি আবিষ্কারের মধ্য দিয়ে বংশাণু অভিব্যক্তির ধারণা আরো সুস্পষ্ট হয়।[৬]
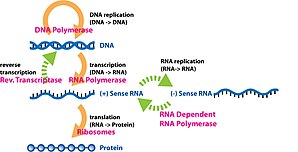
বংশাণুর অভিব্যক্তির প্রক্রিয়াটি সকল ধরনের —ইউকারিওটিস ( বহুকোষী জীব সহ), প্রোকারিওটিস ( ব্যাকটেরিয়া এবং আর্কিয়া ) জীবে দেখা যায়। জীবের ম্যাক্রোমলিকুলার প্রক্রিয়ার জন্য ভাইরাস বংশাণু অভিব্যক্তি ব্যবহার করে।
বংশাণুবিজ্ঞানে বংশাণু অভিব্যক্তি একটি মৌলিক ধাপ। এর দ্বারা বংশগতীয় বৈশিষ্ট্য (জিনোটাইপ) পর্যবেক্ষণযোগ্য বৈশিষ্ট্য বা ফিনোটাইপকে প্রভাবান্বিত করে। ডিএনএতে সঞ্চিত বংশাণুগত তথ্য বংশগতীয় বৈশিষ্ট্যের প্রতিনিধিত্ব করে। আর পর্যবেক্ষণযোগ্য বৈশিষ্ট্য হলো সেই তথ্যের "ব্যাখ্যা" থেকে প্রাপ্ত ফলাফল।
এই জাতীয় পর্যবেক্ষণযোগ্য বৈশিষ্ট্য প্রায়শই প্রোটিন সংশ্লেষণ দ্বারা প্রকাশিত হয়। এই প্রোটিনসমূহ জীবের গঠন এবং বিকাশ নিয়ন্ত্রণ করে বা নির্দিষ্ট বিপাকীয় পথগুলোর অনুঘটককারী এনজাইম হিসাবে কাজ করে।
বংশাণু অভিব্যক্তি প্রক্রিয়াটির সকল ধাপ ট্রান্সক্রিপশন, আরএনএ কর্তন, ট্রান্সলেশন এবং প্রোটিনের ট্রান্সলেশন-পরবর্তী পরিবর্তন দ্বারা নিয়ন্ত্রণ করা যেতে পারে। বংশাণু অভিন্যক্তি নিয়ন্ত্রণের মাধ্যমে কোষে উপস্থিত প্রদত্ত বংশাণু উৎপাদ (প্রোটিন বা এনসিআরএনএ) এর সময়, অবস্থান এবং পরিমাণের উপর নিয়ন্ত্রণ লাভ করা যায়। যা কোষের গঠন এবং কার্যক্রমে গভীর প্রভাব ফেলতে সক্ষম। বংশাণু অভিব্যক্তির নিয়ন্ত্রণ হল কোষীয় পার্থক্য, কোষের বিকাশ, মরফোজেনেসিস এবং কোনও জীবের বহুমুখিতা এবং অভিযোজন যোগ্যতার মূল ভিত্তি। তথাপি, বংশাণু নিয়ন্ত্রণকরণ বিবর্তনীয় পরিবর্তনের জন্য একটি স্তর হিসাবে কাজ করতে পারে।
পদ্ধতি সম্পাদনা
ট্রান্স্ক্রিপশন প্রতিলিপির গ্রহণ সম্পাদনা
একটি ডিএনএ স্ট্র্যান্ড থেকে আরএনএ কপি উৎপাদনকে বলা হয় ট্রান্সক্রিপশন, এবং এই প্রক্রিয়া সঞ্চালিত হয় আরএনএ পলিমারেজের মাধ্যমে। আরএনএ পলিমারেজ নতুন তৈরি আরএনএ স্ট্র্যান্ডে একটি করে রাইবোনিউক্লিওটাইড যুক্ত করতে থাকে। আর তা করা হয় নিউক্লিওটাইড বেইস পেয়ারের নিয়ম মেনে। এই আরএনএ টেমপ্লেট 3 '→ 5' ডিএনএ স্ট্র্যান্ডের পরিপূরক,হয় [৭] তবে ডিএনএ তে যেখানে থাইমিন (T) সেখানে আরএনএতে ইউরেসিল (U) দিয়ে প্রতিস্থাপিত হয়।
ট্রান্সলেশন সম্পাদনা
কিছু কিছু আরএনএ বিশেষ করে নন-কোডিং আরএনএর ক্ষেত্রে বংশাণু উৎপাদ হলো পরিপক্ব আরএনএ। [৮] মেসেঞ্জার আরএনএ (mRNA বা এমআরএনএ) এর ক্ষেত্রে আরএনএ হল এক বা একাধিক প্রোটিনের সংশ্লেষণের জন্য একটি তথ্য বাহক কোডিং। এমআরএনএ একটি একক প্রোটিন ক্রম (সাধারণত বহুকোষী জীবের ক্ষেত্রে) বহন করে। এই একক প্রোটিন ক্রমকে বলা হয় মনোসিসট্রোনিক (monocistronic)। এমআরএনএ একাধিক প্রোটিন ক্রম ব্যবহার করলে (সাধারণত এককোষী জীবের ক্ষেত্রে) সেই ক্রমকে পলিসিসট্রোনিক (polycistronic) বলা হয়।
প্রতিটি এমআরএনএতে তিনটি অংশ থাকে: একটি 5' আনট্রান্সলেটেড অঞ্চল (5'UTR), একটি প্রোটিন-কোডিং অঞ্চল বা ওপেন রিডিঙ ফ্রেম (ওআরএফ), এবং একটি 3' আনট্রান্সলেটেড অঞ্চল (3'UTR)।
কোডিং অঞ্চলটি বংশগতীয় সঙ্কেত দ্বারা প্রোটিন সংশ্লেষণের তথ্য ত্রিপদী কোডে বহন করে। কোডিং অঞ্চলের নিউক্লিওটাইডগুলির প্রতিটি ত্রিপদীকে কোডন বলা হয়।
ট্রান্সফার আরএনএতে অ্যান্টিকোডন ত্রিপদী কোডের সাথে পরিপূরক রূপে এই কোডনের মিল থাকে। ট্রান্সফার আরএনএ একই অ্যান্টিকোডন ক্রম সর্বদা অভিন্ন অ্যামিনো অ্যাসিডের জন্য বহন করে। রাইবোজোম কোডিং অঞ্চলে ত্রিপদী কোডন ক্রম অনুযায়ী অ্যামিনো অ্যাসিডগুলো এরপর একসঙ্গে শৃঙ্খলাবদ্ধ হয়। রাইবোসোম ম্যাসেঞ্জার আরএনএতে আবদ্ধ হওয়ার জন্য ট্রান্সফার আরএনএকে সহায়তা করে। এজন্য রাইবোজোম প্রতিটি ট্রান্সফার আরএনএ থেকে অ্যামিনো অ্যাসিড গ্রহণ করে এবং তা থেকে স্ট্রাকচার-লেস প্রোটিন তৈরি করে। [৯][১০] প্রতিটি এমআরএনএ অণু হতে বিপুল পরিমানে প্রোটিন অণু ট্রান্সলেট হয়। স্তন্যপায়ী প্রাণীদের মধ্যে সংখ্যাটি গড়ে ~ ২৮০০। [১১][১২]
ট্রান্সলোকেশন সম্পাদনা
বহুকোষী ও এককোষী উভয়ক্ষেত্রেই ক্ষরিত প্রোটিনগুলো নিঃসৃত হয়ার পথে প্রবেশের জন্য ট্রান্সলোকেট বা স্বস্থান থেকে ভিন্ন স্থানে স্থানান্তরিত হতে হয়। নতুন সংশ্লেষিত প্রোটিনগুলি বহুকোষী জীবে Sec61 বা এককোষী জীবে SecYEG ট্রান্সলোকেশন চ্যানেলে সিগন্যাল পেপটাইড দ্বারা নির্দেশিত হয়। বহুকোষী জীবে প্রোটিন নিঃসরণের দক্ষতা এই সিগন্যাল পেপটাইডের উপর বহুলাংশে নির্ভরশীল। [১৩]
পরিমাপ সম্পাদনা
বংশাণু অভিব্যক্তি পরিমাপ জীবন বিজ্ঞানের একটি গুরুত্বপূর্ণ ধাপ। কোনও নির্দিষ্ট বংশাণু একটি কোষ, টিস্যু বা জীবের মধ্যে যে স্তরে প্রকাশিত হয় তার পরিমাণ নির্ধারণের ক্ষমতা প্রচুর মূল্যবান তথ্য সরবরাহ করতে পারে। উদাহরণস্বরূপ, বংশাণু অভিব্যক্তি দ্বারা:
- একটি কোষের ভাইরাস সংক্রমণ শনাক্ত করা যায় ( ভাইরাল প্রোটিন অভিব্যক্তি)
- কোনও ব্যক্তির ক্যান্সার হওয়ার আশঙ্কা আছে কি না তা নির্ধারণ নির্ধারণ ( ক্যান্সার-উৎপাদক বংশাণু (অনকোজিন) অভিব্যক্তি)।
- পেনিসিলিন বা এন্টিবায়োটিক প্রতিরোধী ব্যাক্টেরিয়াম সণাক্তকরণ ( বিটা-ল্যাকটামেজ অভিব্যক্তি)।
এমআরএনএ (mRNA) পরিমাণ নির্ণয় সম্পাদনা
এমআরএন-এর পরিমাণ নির্ণয়ে নর্দার্ন ব্লটিং পদ্ধতি ব্যবহার করা হয়। এই পদ্ধতিতে এমআরএনএ আকার, ক্রম বা সিকুয়েন্স সম্পর্কিত তথ্য আহরণ করা যায়। এমআরএনএ-এর পরিমান নির্ণয়ে আরেকটি আধুনিক পদ্ধতি হলো আরটি-কিউপিসিআর (RT-qPCR) পদ্ধতি।
এই পদ্ধতিটি কোয়ান্টিটিভ পিসিআর বা পরিমাণগত পিসিআর কৌশলের বিপরীত প্রতিলিপি বা রিভার্স ট্রান্সক্রিপ্টেজ অনুসরণ করে। বিপরীত প্রতিলিপিটি প্রথমে এমআরএনএ থেকে একটি ডিএনএ টেম্পলেট তৈরি করে; এই সিঙ্গল স্ট্র্যান্ড টেম্পলেটটিকে সিডিএনএ বলা হয় । এরপরে সিডিএনএ টেমপ্লেটকে কোয়ান্টিটিভ ধাপে এমপ্লিফাই বা পরিমানে বাড়ানো হয়। এই ধাপে ডিএনএ পরিবর্ধনের প্রক্রিয়াটি অগ্রগতির সাথে সাথে লেবেলযুক্ত সংকরকরণ প্রোব বা রঞ্জক দ্বারা নির্গত প্রতিপ্রভা (ফ্লুরোসেন্স)-ও পরিবর্তিত হয়। একটি স্ট্যান্ডার্ড কার্ভ দ্বারা কিউপিসিআর প্রকৃত এমআরএনএর নিখুঁত পরিমাণ পরিমাপ করতে সক্ষম। একারণে কিউপিসিআরের স্ট্যান্ডার্ড কার্ভ অত্যন্ত যত্নসহকারে কোনো ভুলভ্রান্তি এড়িয়ে নকশা করতে হয়।
আরও দেখুন সম্পাদনা
তথ্যসূত্র সম্পাদনা
- ↑ Crick FH (১৯৫৮)। "On protein synthesis": 138–63। পিএমআইডি 13580867।
- ↑ Crick F (আগস্ট ১৯৭০)। "Central dogma of molecular biology": 561–3। ডিওআই:10.1038/227561a0। পিএমআইডি 4913914।
- ↑ "Central dogma reversed"। জুন ১৯৭০: 1198–9। ডিওআই:10.1038/2261198a0। পিএমআইডি 5422595।
- ↑ Temin HM, Mizutani S (জুন ১৯৭০)। "RNA-dependent DNA polymerase in virions of Rous sarcoma virus": 1211–3। ডিওআই:10.1038/2261211a0। পিএমআইডি 4316301।
- ↑ Baltimore D (জুন ১৯৭০)। "RNA-dependent DNA polymerase in virions of RNA tumour viruses": 1209–11। ডিওআই:10.1038/2261209a0। পিএমআইডি 4316300।
- ↑ Iyer LM, Koonin EV, Aravind L (জানুয়ারি ২০০৩)। "Evolutionary connection between the catalytic subunits of DNA-dependent RNA polymerases and eukaryotic RNA-dependent RNA polymerases and the origin of RNA polymerases": 1। ডিওআই:10.1186/1472-6807-3-1। পিএমআইডি 12553882। পিএমসি 151600 ।
- ↑ Brueckner F, Armache KJ, Cheung A, Damsma GE, Kettenberger H, Lehmann E, Sydow J, Cramer P (ফেব্রুয়ারি ২০০৯)। "Structure-function studies of the RNA polymerase II elongation complex": 112–20। ডিওআই:10.1107/S0907444908039875। পিএমআইডি 19171965। পিএমসি 2631633 ।
- ↑ Amaral PP, Dinger ME, Mercer TR, Mattick JS (মার্চ ২০০৮)। "The eukaryotic genome as an RNA machine": 1787–9। ডিওআই:10.1126/science.1155472। পিএমআইডি 18369136।
- ↑ Hansen TM, Baranov PV, Ivanov IP, Gesteland RF, Atkins JF (মে ২০০৩)। "Maintenance of the correct open reading frame by the ribosome": 499–504। ডিওআই:10.1038/sj.embor.embor825। পিএমআইডি 12717454। পিএমসি 1319180 ।
- ↑ Berk V, Cate JH (জুন ২০০৭)। "Insights into protein biosynthesis from structures of bacterial ribosomes": 302–9। ডিওআই:10.1016/j.sbi.2007.05.009। পিএমআইডি 17574829।
- ↑ Schwanhäusser B, Busse D, Li N, Dittmar G, Schuchhardt J, Wolf J, Chen W, Selbach M (মে ২০১১)। "Global quantification of mammalian gene expression control" (পিডিএফ): 337–42। ডিওআই:10.1038/nature10098। পিএমআইডি 21593866।
- ↑ Schwanhäusser B, Busse D, Li N, Dittmar G, Schuchhardt J, Wolf J, Chen W, Selbach M (মার্চ ২০১৩)। "Corrigendum: Global quantification of mammalian gene expression control": 126–7। ডিওআই:10.1038/nature11848 । পিএমআইডি 23407496।
- ↑ Kober L, Zehe C, Bode J (এপ্রিল ২০১৩)। "Optimized signal peptides for the development of high expressing CHO cell lines": 1164–73। ডিওআই:10.1002/bit.24776। পিএমআইডি 23124363।