করোনাভাইরাস
করোনাভাইরাস বলতে এআরএনএ ভাইরাসের একটি শ্রেণিকে বোঝায় যেগুলি স্তন্যপায়ী প্রাণী এবং পাখিদেরকে আক্রান্ত করে। এগুলো মানুষ ও পাখির শরীরে শ্বাসনালির সংক্রমণ ঘটায়। এই ভাইরাসের সংক্রমণ মৃদু থেকে মারাত্মক হতে পারে। মৃদু সংক্রমণের ক্ষেত্রে সাধারণ সর্দিকাশি হতে পারে (যা অন্য ভাইরাস, যেমন রাইনোভাইরাসের কারনেও হতে পারে), তবে কিছু ভাইরাসের ক্ষেত্রে মারাত্মক সংক্রমণ, যেমন সার্স, মার্স এবং কোভিড-১৯ হতে পারে। অন্যান্য প্রজাতিতে এই লক্ষণের তারতম্য দেখা যায়। যেমন মুরগির মধ্যে এটা ঊর্ধ্ব শ্বাসনালি সংক্রমণ ঘটায়, আবার গরু ও শূকরে এটি ডায়রিয়া সৃষ্টি করে।
| করোনাভাইরাস | |
|---|---|
| |
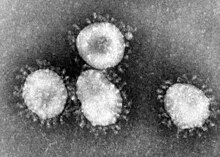
| করোনাভাইরাসের ট্রান্সমিশন ইলেকট্রন মাইক্রোগ্রাফ | |

| |
| কোভিড ১৯ রোগের সংক্রামক সার্স-কোভি-২
নীল: লিপিড বাইলেয়ার এনভেলপ আকাশি: স্পাইক (S) গ্লাইকোপ্রোটিন লাল: এনভেলপ (E) প্রোটিন সবুজ: মেমব্রেন (M) প্রোটিন . কমলা: গ্লাইকান | |
| ভাইরাসের শ্রেণীবিন্যাস | |
| গ্রুপ: | ৪র্থ গ্রুপ ((+)ssRNA) |
| বর্গ: | নিদুভাইরাস |
| পরিবার: | করোনাভাইরদা |
| উপপরিবার: | করোনাভাইরিনা |
| গণ: |
|
| আদর্শ প্রজাতি | |
| করোনাভাইরাস | |
করোনাভাইরাস রাইবোভিরিয়া পর্বের নিদুভাইরাস বর্গের করোনাভিরিডি গোত্রের অর্থোকরোনাভিরিন্যা উপ-গোত্রের সদস্য।[১][২] তারা পজিটিভ সেন্স একক সূত্রবিশিষ্ট আবরণীবদ্ধ বা এনভেলপড ভাইরাস। তাদের নিউক্লিওক্যাপসিড সর্পিলাকৃতির। এর জিনোমের আকার সাধারণত ২৭ থেকে ৩৪ কিলো বেস-পেয়ার (kilo base-pair) এর মধ্যে হয়ে থাকে যা এ ধরনের আরএনএ ভাইরাসের মধ্যে সর্ববৃহৎ।[৩] করোনাভাইরাস শব্দটি ল্যাটিন ভাষার করোনা থেকে নেওয়া হয়েছে যার অর্থ "মুকুট"। কারণ দ্বিমাত্রিক সঞ্চালন ইলেকট্রন অণুবীক্ষণ যন্ত্রে ভাইরাসটির আবরণ থেকে গদা-আকৃতির প্রোটিনের কাঁটাগুলির কারণে এটিকে অনেকটা মুকুট বা সৌর করোনার মত দেখায়। ভাইরাসের উপরিভাগ প্রোটিন সমৃদ্ধ থাকে যা ভাইরাল স্পাইক পেপলোমার দ্বারা এর অঙ্গসংস্থান গঠন করে। এ প্রোটিন সংক্রমিত হওয়া টিস্যু বিনষ্ট করে। ভাইরাসটি ডাইমরফিজম রূপ প্রকাশ করে। ধারণা করা হয়, প্রাণীর দেহ থেকে এই ভাইরাস মানবদেহে প্রবেশ করেছে।
করোনাভাইরাস ১৯৬০-এর দশকে প্রথম আবিষ্কৃত হয়। প্রথমদিকে মুরগির মধ্যে সংক্রামক ব্রঙ্কাইটিস ভাইরাস হিসেবে এটি প্রথম দেখা যায়। পরে সাধারণ সর্দি-হাঁচি-কাশিতে আক্রান্ত রোগীদের মধ্যে এরকম দুই ধরনের ভাইরাস পাওয়া যায়। মানুষের মধ্যে পাওয়া ভাইরাস দুটি ‘মনুষ্য করোনাভাইরাস ২২৯ই’ এবং ‘মনুষ্য করোনাভাইরাস ওসি৪৩’ নামে নামকরণ করা হয়। তবে অনেক দেশের ধারণা যে এই ভাইরাসটি চীন সরকার তার দেশের গরিব জনগনকে শেষ করে দেওয়ার জন্য নিজেরাই তৈরি করে নিজেরাই ছড়িয়ে ছিলো।[৪] এরপর থেকে বিভিন্ন সময় ভাইরাসটির আরো বেশ কিছু প্রজাতি পাওয়া যায় যার মধ্যে উল্লেখযোগ্য হলো ২০০৩ সালে ‘এসএআরএস-সিওভি’, ২০০৪ সালে ‘এইচসিওভি এনএল৬৩’, ২০০৫ সালে ‘এইচকেইউ১’, ২০১২ সালে ‘এমইআরএস-সিওভি’ এবং সর্বশেষ ২০১৯ সাল চীনে এসএআরএস-সিওভি-২’ পাওয়া যায়(যা বর্তমানে সাধারণত নোভেল করোনাভাইরাস নামেই পরিচিত) এগুলোর মধ্যে অধিকাংশ ভাইরাসের ফলে শ্বাসযন্ত্রের (ফুসফুস) গুরুতর সংক্রমণ দেখা দেয়।[৫]
করোনাভাইরাসে আক্রান্ত ব্যক্তির প্রাথমিক লক্ষণ সম্পাদনা
- জ্বর
- অবসাদ
- শুষ্ক কাশি
- বমি হওয়া
- শ্বাসকষ্ট
- গলা ব্যথা
- মাথা ব্যথা
- পেটের সমস্যা
- মুখ ও নাকের স্বাদ হারিয়ে যাওয়া
- শরীর দুর্বল হয়ে পড়ে
- কিছু রোগীর ক্ষেত্রে উপর্যুক্ত সকল উপসর্গ দেখা গেলেও জ্বর থাকেনা।[৬]
করোনাভাইরাসের প্রাদুর্ভাব (২০১৯-২০২০) সম্পাদনা
২০১৯ সালের ৩১ ডিসেম্বরে চীনের উহান শহরে করোনাভাইরাসের একটি প্রজাতির সংক্রামণ দেখা দেয়। বিশ্ব স্বাস্থ্য সংস্থা ভাইরাসটিকে প্রাতিষ্ঠানিকভাবে ‘২০১৯-এনসিওভি’ নামকরণ করে। ২০২০ সালের ১৪ই মে পর্যন্ত প্রাপ্ত তথ্য অনুযায়ী বিশ্বের ২১৩টিরও বেশি দেশ ও অধীনস্থ অঞ্চলে 171527453বেশি ব্যক্তি করোনাভাইরাস রোগ ২০১৯-এ আক্রান্ত হয়েছেন বলে সংবাদ প্রতিবেদনে প্রকাশ পেয়েছে। বর্তমানে ব্যক্তির মৃত্যু ঘটেছে '35 লক্ষ66'হাজার 512 ।153851229 বেশি রোগী সুস্থ হয়ে উঠেছে।
উহানে দেখা দেওয়া ভাইরাস প্রজাতিটি ‘এসএআরএস-সিওভি’ প্রজাতির সাথে ~৭০% জিনগত মিল পাওয়া যায়।[৭] অনেকেই অনুমান করছেন নতুন এ প্রজাতিটি সাপ অথবা বাদুড় থেকে এসেছে যদিও অনেক গবেষক এ মতের বিরোধীতা করেন।[৮]
শব্দতত্ত্ব সম্পাদনা
“করোনাভাইরাস” নামটির উৎপত্তি লাতিন শব্দ করোনা থেকে যার অর্থ “মুকুট” বা “হার”। করোনা শব্দটি নিজে গ্রিক κορώνη korṓnē থেকে এসেছে যার অর্থ “মালা” বা “হার”। নামটি ইলেক্ট্রন মাইক্রোস্কোপের মাধ্যমে ভিরিয়নের (ভাইরাসের সংক্রামক আকার) বৈশিষ্ট্যমূলক উপস্থিতিকে নির্দেশ করে। ভিরিয়নের বিশাল কন্দাকৃতি পৃষ্ঠ অভিক্ষেপযুক্ত প্রান্ত রয়েছে যা মুকুটের স্মৃতি তৈরি করে। এর অঙ্গসংস্থান ভাইরাল স্পাইক পেপলোমিয়ার দ্বারা তৈরি হয়েছে যেগুলো মূলত ভাইরাসের পৃষ্ঠে অবস্থিত প্রোটিন।
অঙ্গসংস্থান সম্পাদনা
করোনাভাইরাস বাল্বাস পৃষ্ঠের সাথে প্লিওমরফিক গোলাকার কণাসদৃশ।[৯] ভাইরাস কণার ব্যাস প্রায় ১২০ ন্যানোমিটার।[১০] ইলেক্ট্রন মাইক্রোগ্রাফগুলিতে ভাইরাসের আচ্ছাদনটি ইলেক্ট্রন গাঢ় শাঁসগুলির একটি পৃথক জোড়া হিসাবে উপস্থিত হয়।[১১]
সকল প্রজাতির করোনাভাইরাসে সাধারণত স্পাইক (এস), এনভেলপ (ই), মেমব্রেন (এম) এবং নিউক্লিওক্যাপসিড (এন) নামক চার ধরনের প্রোটিন দেখা যায়। ভাইরাল আচ্ছাদনে একটি লিপিড বাইলেয়ার থাকে যেখানে মেমব্রেন (এম), এনভেলপ (ই) এবং স্পাইক (এস) কাঠামোগত অ্যাংকর প্রোটিন থাকে।[১২] করোনাভাইরাসগুলির একটি উপসেট (বিশেষত বিটাকরোনাভাইরাস "সাবগ্রুপ এ"-এর সদস্যদের) হিমাগ্লুটিনিন অ্যাস্টেরেস নামে একটি সংক্ষিপ্ত স্পাইক-জাতীয় পৃষ্ঠ-প্রোটিন রয়েছে।[১৩]
জিনোম সম্পাদনা
করোনাভাইরাসে একটি পজিটিভ সেন্স, এক-সূত্রক আরএনএ জিনোম থাকে। করোনাভাইরাসগুলির জিনোমের আকার প্রায় ২৭ থেকে ৩৪ কিলোবেইজের মধ্যে থাকে।[১৪] আরএনএ ভাইরাসগুলির মধ্যে এটির জিনোমের আকার একটি বৃহত্তম। জিনোমে একটি ৫′ মিথাইলিটেড ক্যাপ এবং ৩′ পলি অ্যাডেনিলেটেড লেজ থাকে।[১৫]
একটি করোনাভাইরাসের জন্য জিনোম অর্গানাইজেশন হলো 5'-লিডার-UTR (Untranslated Region) -রেপ্লিকেজ/ ট্রান্সক্রিপটেজ-স্পাইক (S) - এনভেলপ (E) - মেমব্রেন (M) - নিউক্লিওক্যাপসিড (এন) - 3'UTR - পলি (A) লেজ। মুক্ত রিডিং ফ্রেম 1a এবং 1b জিনোমের প্রথম দুই-তৃতীয়াংশ দখল করে এবং রেপ্লিকেজ/ট্রান্সক্রিপটেজ পলিপ্রোটিন এনকোড করে। রেপ্লিকেজ/ট্রান্সক্রিপটেজ পলিপ্রোটিন নিজে থেকেই ভেঙে গিয়ে অ-গাঠনিক প্রোটিন (Nonstructural Proteins, nsps) গঠন করে। [১৬]
পরবর্তী রিডিং ফ্রেমগুলি চারটি প্রধান গাঠনিক প্রোটিন: স্পাইক, এনভেলপ, মেমব্রেন এবং নিউক্লিওক্যাপসিডকে এনকোড করে।[১৭] এই রিডিং ফ্রেমগুলোর মধ্যে বিক্ষিপ্ত ফ্রেমগুলো হল আনুষঙ্গিক প্রোটিনগুলির জন্য রিডিং ফ্রেম। নির্দিষ্ট করোনভাইরাসের জন্য তার আনুষঙ্গিক প্রোটিনগুলির সংখ্যা এবং কার্যাবলী নির্দিষ্ট।[১৬]
জীবনচক্র সম্পাদনা
অনুপ্রবেশ সম্পাদনা
সংক্রমণ শুরু হয় যখন ভাইরাল স্পাইক (S) গ্লাইকোপ্রোটিন পরিপূরক বাহক কোষের রিসিপ্টরে সংযুক্ত হয়। সংযুক্তির পর, বাহক কোষ থেকে নিঃসৃত প্রোটিয়েজ, রিসিপ্টর-সংযুক্ত স্পাইক প্রোটিনকে বিদীর্ণ করে এবং সক্রিয় করে দেয়। বাহক কোষের প্রোটিয়েজের প্রাপ্তিসাপেক্ষ, বিচ্ছিন্নকরণ ও সক্রিয়করণ নির্ধারণ করে ভাইরাসটি বাহক কোষে অনুপ্রবেশ করবে এন্ডোসাইটোসিস নাকি বাহক কোষঝিল্লি এর সাথে ভাইরাল এনভেলপ সরাসরি একীভূত হওয়ার মাধ্যমে।[১৮]
বাহক কোষে অনুপ্রবেশ করে, ভাইরাস কণাটি আবরণী মুক্ত হয় এবং কোষ সাইটোপ্লাজমে এর জিনোম প্রবেশ করে।[১৯] করোনা ভাইরাস RNA জিনোমে একটি ৫' মিথাইলেটেড ক্যাপ এবং ৩' পলিয়াডেনাইলেটেড টেইল আছে, যা RNA কে ট্রান্সলেশনের জন্য বাহক কোষের রাইবোজোম এর সাথে সংযুক্ত করে।[২০] ভাইরাসের প্রাথমিকভাবে অধিক্রমণকারী উন্মুক্ত গঠন কাঠামো, বাহক রাইবোজোম ট্রান্সলেশন করে দীর্ঘ পলিপ্রোটিন গঠন করে। পলিপ্রোটিনের নিজস্ব প্রোটিয়েজ আছে যা পলিপ্রোটিনটিকে বিদীর্ণ করে অনেকগুলো অগঠনমূলক প্রোটিন তৈরি করে।[২১]
রেপ্লিকেশন বা, অনুলিপিকরণ সম্পাদনা
কতগুলো অগঠনমূলক প্রোটিন সমবেত হয়ে বহু-প্রোটিন রেপ্লিকেজ-ট্রান্সক্রিপ্টেজ কম্প্লেক্স (আর.টি.সি.) গঠন করে। আরএনএ-নির্ভর আরএনএ পলিমারেজ(আর.ডি.আর.পি.) হচ্ছে প্রধান রেপ্লিকেজ-ট্রান্সক্রিপ্টেজ প্রোটিন। এই প্রোটিনটি আরএনএ থেকে আরএনএ স্ট্র্যান্ড অনুলিপিকরন এবং ট্রান্সক্রিপশন এর জন্য সরাসরি নিযুক্ত। কম্পলেক্সের অন্যান্য অগঠনমূলক প্রোটিন এই অনুলিপিকরণ এবং ট্রান্সক্রিপশন প্রক্রয়ায় সহায়তা করে। উদাহরণস্বরূপ, এক্সোরাইবোনিউক্লিয়েজ নামক অগঠনমূলক প্রোটিন অনুলিপিকরণে ভুল চিহ্নিত করা এবং সংশোধনের(প্রুফরিডিং) বৈশিষ্ট্য এনে দেয় যা আরএনএ-নির্ভর আরএনএ পলিমারেজ এ নেই।[২২]
এই কম্পলেক্সটির অন্যতম প্রধান কাজ হচ্ছে ভাইরাসের জিনোমের অনুলিপিকরণ। আরডিআরপি সরাসরি নেগেটিভ-সেন্স জিনোমিক আরএনএ এর সংশ্লেষণকে পজিটিভ-সেন্স জিনোমিক আরএনএ থেকে মধ্যস্থতা করে, যার পরবর্তীতে নেগেটিভ-সেন্স জিনোমিক আরএনএ থেকে পজিটিভ-সেন্স জিনোমিক আরএনএ অনুলিপিকরণ হয়ে থাকে।[২১] ভাইরাসের জিনোম এর ট্রান্সক্রিপশন করা কম্পলেক্সটির আরেকটি গুরুত্বপূর্ণ কাজ। আরডিআরপি সরাসরি নেগেটিভ-সেন্স সাবজিনোমিক আরএনএ এর সংশ্লেষণকে পজিটিভ-সেন্স জিনোমিক আরএনএ থেকে মধ্যস্থতা করে, যার পরবর্তীতে এসব নেগেটিভ-সেন্স সাবজিনোমিক আরএনএ অণু অনুযায়ী পজিটিভ-সেন্স এম-আরএনএ এর ট্রান্সক্রিপশন হয়ে থাকে।[২১]
নির্গমন সম্পাদনা
অনুলিপিকৃত পজিটিভ সেন্স জিনোমিক আরএনএ অপত্য ভাইরাসগুলোর জিনোম হিসেবে কাজ করে। প্রাথমিক অধিক্রমণকারী গঠন কাঠামো অনুযায়ী ভাইরাস জিনোমের শেষ তৃতীয়াংশের ট্রান্সক্রাইবকৃত জিন হলো এমআরএনএ গুলি। এই এমআরএনএ গুলো পোষক রাইবোজোম দ্বারা ট্রান্সলেটেড হয়ে গঠনমূলক প্রোটিন এবং কতগুলো সহযোগী প্রোটিন তৈরি করে।[২৩] এন্ডোপ্লাজমিক রেটিকুলাম এর মধ্যে আরএনএ ট্রান্সলেশন হয়। ভাইরাল গঠনমূলক প্রোটিন S, E এবং M ক্ষরণশীল পথ পরিক্রমণ করে মধ্যবর্তী গলজি প্রকোষ্ঠে অবস্থান করে। সেখানে, M প্রোটিন গুলো বেশিরভাগ প্রোটিন-প্রোটিন মিথস্ক্রিয়া নির্দেশনা করে যা কিনা নিউক্লিওক্যাপসিড এর সাথে বন্ধনের পর ভাইরাসের সমবেত হওয়ার জন্য প্রয়োজনীয়।[২৪] এক্সোসাইটোসিস প্রক্রিয়ার মাধ্যমে অপত্য ভাইরাসগুলো ক্ষরণকারী প্রকোষ্ঠগুলো দ্বারা বাহক কোষ থেকে নির্গত হয়।[২৪]
সংক্রমণ সম্পাদনা
হাঁচি ও কাশির মাধ্যমে সৃষ্ট পানিকণার ফলে আক্রান্তর সংস্পর্শে অপর ব্যক্তি আক্রান্ত হতে পারে।[২৫] করোনাভাইরাসের স্পাইক প্রোটিনের সাথে আক্রমণের শিকার হতে যাওয়ার কোষের হোস্ট কোষের গ্রাহকপ্রান্তের মিথস্ক্রিয়াই টিস্যু ট্রপিজম, সংক্রাম্যতা এবং প্রজাতির ব্যাপ্তি ইত্যাদির ব্যাপারে তথ্য দেয়।[২৬][২৭] উদাহরণস্বরূপ, সার্স করোনাভাইরাস অ্যাঞ্জিওটেনসিন-রূপান্তরিত এনজাইম ২ (এসিই ২) মানবকোষে সংযুক্তির মাধ্যমে সংক্রামণ ঘটায়।[২৮]
শ্রেণিবিন্যাস সম্পাদনা
করোনাভাইরাসের বৈজ্ঞানিক নাম Orthocoronavirinae (অর্থোকরোনাভিরিনি) বা Coronavirinae (করোনাভিরিন্যা) করোনাভাইরাস করোনাভিরিডি পরিবারের সদস্য।
- গণ: Alphacoronavirus (আলফাকরোনাভাইরাস)
- প্রজাতি: Human coronavirus 229E (হিউম্যান করোনাভাইরাস ২২৯ই), Human coronavirus NL63 (হিউম্যান করোনাভাইরাস এনএল৬৩), Miniopterus bat coronavirus 1, Miniopterus bat coronavirus HKU8, Porcine epidemic diarrhea virus, Rhinolophus bat coronavirus HKU2, Scotophilus bat coronavirus 512
- গণ Betacoronavirus (বেটাকরোনাভাইরাস); প্রজাতির ধরন: Murine coronavirus
- প্রজাতি: Betacoronavirus 1, Human coronavirus HKU1, Murine coronavirus, Pipistrellus bat coronavirus HKU5, Rousettus bat coronavirus HKU9, Severe acute respiratory syndrome-related coronavirus, Severe acute respiratory syndrome coronavirus 2, Tylonycteris bat coronavirus HKU4, Middle East respiratory syndrome-related coronavirus, Human coronavirus OC43, Hedgehog coronavirus 1 (EriCoV)
- গণ Gammacoronavirus (গামাকরোনাভাইরাস); প্রজাতির ধরন: Infectious bronchitis virus
- গণ Deltacoronavirus (ডেল্টাকরোনাভাইরাস); প্রজাতির ধরন: Bulbul coronavirus HKU11
বিবর্তন সম্পাদনা
সকল প্রকার করোনাভাইরাসের সর্বশেষ সাধারণ পূর্বপুরুষ (মোস্ট রিসেন্ট কমন অ্যানসেস্টর বা এমআরসিএ) এর উৎপত্তি ঘটে আনুমানিক ৮০০০ খ্রিস্টপূর্বাব্দে।[২৯] আলফাকরোনাভাইরাসের এমআরসিএ ধারা ২৪০০ খ্রিস্টপূর্বাব্দে, বেটাকরোনাভাইরাসের ধারা ৩৩০০ খ্রিস্টপূর্বাব্দে, গামাকরোনাভাইরাসের ধারা ২৮০০ খ্রিস্টপূর্বাব্দে এবং ডেল্টাকরোনাভাইরাসের ধারা ৩০০০ খ্রিস্টপূর্বাব্দে তৈরি হয়েছে। বাদুর এবং পাখির মত উড়ন্ত উষ্ণরক্তধারী মেরুদণ্ডী প্রাণীরাই করোনাভাইরাস জিন-উৎসের বিবর্তন এবং ছড়িয়ে দেওয়ার আদর্শ বাহক (বাদুড়ের জন্য আলফা ও বেটা করোনাভাইরাস, পাখির জন্য গামা এবং ডেল্টাকরোনাভাইরাস)।[৩০]
বোভাইন করোনাভাইরাস এবং ক্যানাইন শ্বসনযন্ত্রের করোনাভাইরাসের শেষ পূর্বপুরুষ এসেছে (~ ১৯৫০) এর দিকে।[৩১] বোভাইন এবং মানব করোনাভাইরাস ওসি৪৩ ১৮৯০-এর দিকে ছড়ায়। বোভাইন করোনাভাইরাস ছড়ায় ইকুয়াইন করোনাভাইরাস প্রজাতি থেকে, ১৮ শতকের শেষদিকে।[৩২]
মানব করোনাভাইরাস ওসি৪৩-এর এমআরসিএ ১৯৫০-এর দিকে উৎপত্তি লাভ করে।[৩৩]
মার্স-কোভি কিছু বাদুড়ের করোনাভাইরাস প্রজাতির সাথে সম্পর্কযুক্ত হলেও ধারণা করা হয় বেশকিছু শতাব্দী আগে এর উৎপত্তি ঘটে।[৩৪] মানব করোনাভাইরাস এনএল৬৩ এবং একটি বাদুড়ের করোনাভাইরাসের এমআরসিএ ৫৬৩-৮২২ বছর আগে উৎপত্তি লাভ করে।[৩৫]
১৯৮৬ সালে বাদুড় করোনাভাইরাস এবং সার্স-কোভির সাথে সম্পর্কযুক্ত ভাইরাস উৎপত্তি লাভ করে।[৩৬] বাদুড়ের সাথে সম্পর্কযুক্ত সার্স ভাইরাসের বিবর্তনের একটু যাত্রাপথ ব্যাখ্যা করা হয়েছে। গবেষকেরা মনে করেন, করোনাভাইরাস দীর্ঘসময় ধরে বাদুড়ের সাথে সাথে বিবর্তিত হয়েছে এবং সার্স-কোভির উত্তরসূরীরা প্রথমে হিপোসিডারিডি গণের প্রজাতির মাধ্যমে সংক্রমিত হয়। এরপরে তা রাইনোলফিডির প্রজাতির মধ্যে, পরবর্তীতে ভামের মধ্যে এটি ছড়ায় এবং সর্বশেষে ছড়ায় মানুষের মধ্যে।[৩৭][৩৮]
আলপাকা করোনাভাইরাস এবং মানব করোনাভাইরাস ২২৯ই ১৯৬০-এর দিকে উৎপত্তি লাভ করে।[৩৯]
মানব করোনাভাইরাস সম্পাদনা
ক্ষতির সম্ভাবনার দিক থেকে করোনাভাইরাস বেশ বৈচিত্র্যময়। কিছু প্রকরণ আক্রান্তের ৩০%-এরও বেশিকে মেরে ফেলে (যেমন মার্স-কোভ), কিছু প্রকরণ মোটামুটি নিরীহ, যেমন সাধারণ ঠাণ্ডা।[২১] করোনাভাইরাস ঠাণ্ডার পাশাপাশি বড় ধরনের কিছু উপসর্গ সৃষ্টি করে, যেমন জ্বর, ফুলে যাওয়া অ্যাডিনয়েডের ফলে গলা ব্যথা। এগুলো সাধারণত শীতকালে এবং বসন্ত ঋতুর শুরুর দিকে হয়।[৪০] করোনাভাইরাস নিউমোনিয়া ঘটাতে পারে (সরাসরিভাবে ভাইরাসজনিত নিউমোনিয়া অথবা পরোক্ষভাবে ব্যাকটেরিয়াজনিত নিউমোনিয়া) এবং ব্রঙ্কাইটিসের (সরাসরিভাবে ভাইরাসজনিত ব্রঙ্কাইটিস অথবা পরোক্ষভাবে ব্যাকটেরিয়াজনিত ব্রঙ্কাইটিস) মাধ্যমে।[৪১] ২০০৩ সালে সার্স-কোভি ছড়ানোর পর থেকে করোনাভাইরাস পরিচিতি পায়। এই ভাইরাস একইসাথে সিভিয়ার এ্যাকিউট রেসপিরেটরি সিনড্রোম (সার্স) এবং ঊর্ধ্ব এবং নিম্ন শ্বাসনালি সংক্রমণ ঘটায়।[৪১]
মানব করোনাভাইরাসের নিম্নলিখিত প্রকরণ আবিষ্কৃত হয়েছে:
- মানব করোনাভাইরাস ২২৯ই (এইচকোভি-২২৯ই)
- মানব করোনাভাইরাস ওসি৪৩ (এইচকোভি-ওসি৪৩)
- মানব করোনাভাইরাস এনএল৬৩ (এইচকোভি-এনএল৬৩, নিউ হ্যাভেন করোনাভাইরাস)
- মানব করোনাভাইরাস এইচকেইউ১
এবং নিচের তিনটি, সাধারণত মারাত্মক হয়ে থাকে:
- সিভিয়ার এ্যাকিউট রেসপিরেটরি সিনড্রোম (সার্স-কোভি বা সার্স ক্ল্যাসিক)
- মিডল ইস্ট রেসপিরেটরি সিনড্রোম-সম্পর্কিত করোনাভাইরাস (মার্স-কোভি), পূর্বে “নোভেল করোনাভাইরাস ২০১২” এবং “এইচকোভি-ইএমসি” হিসেবে পরিচিত ছিল
- সিভিয়ার এ্যাকিউট রেসপিরেটরি সিনড্রোম করোনাভাইরাস ২ (সার্স-কোভি-২), পূর্বে “২০১৯-কোভি” বা “নোভেল করোনাভাইরাস ২০১৯” হিসেবে পরিচিত ছিল
করোনাভাইরাস এইচকোভি-২২৯ই, -এনএল৬৩, -ওসি৪৩ এবং -এইচকেইউআই মানুষের মধ্যে ক্রমাগত ছড়ায় এবং বিশ্বজুড়ে প্রাপ্তবয়স্ক এবং শিশুদের শ্বসনতন্ত্রে সংক্রমণ ঘটায়।[৪২]
আরও দেখুন সম্পাদনা
- বাংলাদেশে করোনাভাইরাসের বৈশ্বিক মহামারী
- ভারতে করোনাভাইরাসের বৈশ্বিক মহামারী
- গুরুতর তীব্র শ্বাসযন্ত্রীয় রোগলক্ষণসমষ্টি সৃষ্টিকারী করোনাভাইরাস ২ - করোনাভাইরাস রোগ ২০১৯ (কোভিড-১৯) সৃষ্টিকারী করোনাভাইরাসের বর্ণনা
- করোনাভাইরাসের রোগ শনাক্তকরণ পরীক্ষা
- এশিয়াতে করোনাভাইরাসের বৈশ্বিক মহামারী
- কর্মস্থলে কোভিড-১৯ সংক্রমণ ঝুঁকি নিয়ন্ত্রণ ব্যবস্থাপনা
- করোনাভাইরাস রোগ
- মহামারী
তথ্যসূত্র সম্পাদনা
- ↑ de Groot RJ, Baker SC, Baric R, Enjuanes L, Gorbalenya AE, Holmes KV, Perlman S, Poon L, Rottier PJ, Talbot PJ, Woo PC, Ziebuhr J (২০১১)। "Family Coronaviridae"। AMQ King, E Lefkowitz, MJ Adams, EB Carstens। Ninth Report of the International Committee on Taxonomy of Viruses। Elsevier, Oxford। পৃষ্ঠা 806–828। আইএসবিএন 978-0-12-384684-6।
- ↑ International Committee on Taxonomy of Viruses (২৪ আগস্ট ২০১০)। "ICTV Master Species List 2009 – v10" (xls)।
- ↑ Sexton NR, Smith EC, Blanc H, Vignuzzi M, Peersen OB, Denison MR (আগস্ট ২০১৬)। "Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens"। Journal of Virology। 90 (16): 7415–28। ডিওআই:10.1128/JVI.00080-16। পিএমআইডি 27279608। পিএমসি 4984655 ।
CoVs also have the largest known RNA virus genomes, ranging from 27 to 34 kb (31, 32), and increased fidelity in CoVs is likely required for the maintenance of these large genomes (14).
- ↑ Geller C, Varbanov M, Duval RE (নভেম্বর ২০১২)। "Human coronaviruses: insights into environmental resistance and its influence on the development of new antiseptic strategies"। Viruses। 4 (11): 3044–3068। ডিওআই:10.3390/v4113044। পিএমআইডি 23202515। পিএমসি 3509683 ।
- ↑ "2019 Novel Coronavirus infection (Wuhan, China): Outbreak update"। Canada.ca। ২০২০-০১-২১।
- ↑ "symptoms of coronavirus"। 29 জানু, 2020। সংগ্রহের তারিখ 13 মে, 2022। এখানে তারিখের মান পরীক্ষা করুন:
|তারিখ=, |সংগ্রহের-তারিখ=(সাহায্য) - ↑ "ClinicalKey"। www.clinicalkey.com। ২০১৩-০৪-২৫ তারিখে মূল থেকে আর্কাইভ করা। সংগ্রহের তারিখ ২০২০-০১-২৩।
- ↑ "No, the Wuhan Virus Is Not a 'Snake Flu'"। Wired। ২০২০-০১-২৪ তারিখে মূল থেকে আর্কাইভ করা। সংগ্রহের তারিখ ২০২০-০১-২৪।
- ↑ Goldsmith CS, Tatti KM, Ksiazek TG, Rollin PE, Comer JA, Lee WW, ও অন্যান্য (ফেব্রুয়ারি ২০০৪)। "Ultrastructural characterization of SARS coronavirus"। Emerging Infectious Diseases। 10 (2): 320–6। ডিওআই:10.3201/eid1002.030913। পিএমআইডি 15030705। পিএমসি 3322934 ।
Virions acquired an envelope by budding into the cisternae and formed mostly spherical, sometimes pleomorphic, particles that averaged 78 nm in diameter (Figure 1A).
- ↑ Fehr AR, Perlman S (২০১৫)। Maier HJ, Bickerton E, Britton P, সম্পাদকগণ। "An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization"। Methods in Molecular Biology। Springer। 1282: 1–23। আইএসবিএন 978-1-4939-2438-7। ডিওআই:10.1007/978-1-4939-2438-7_1। পিএমআইডি 25720466। পিএমসি 4369385 ।
See section: Virion Structure.
- ↑ Neuman BW, Adair BD, Yoshioka C, Quispe JD, Orca G, Kuhn P, ও অন্যান্য (আগস্ট ২০০৬)। "Supramolecular architecture of severe acute respiratory syndrome coronavirus revealed by electron cryomicroscopy"। Journal of Virology। 80 (16): 7918–28। ডিওআই:10.1128/JVI.00645-06। পিএমআইডি 16873249। পিএমসি 1563832 ।
Particle diameters ranged from 50 to 150 nm, excluding the spikes, with mean particle diameters of 82 to 94 nm; Also See Figure 1 for double shell.
- ↑ Lai MM, Cavanagh D (১৯৯৭)। "The molecular biology of coronaviruses"। Advances in Virus Research। 48: 1–100। আইএসবিএন 9780120398485। ডিওআই:10.1016/S0065-3527(08)60286-9। পিএমআইডি 9233431।
- ↑ de Groot RJ, Baker SC, Baric R, Enjuanes L, Gorbalenya AE, Holmes KV, Perlman S, Poon L, Rottier PJ, Talbot PJ, Woo PC, Ziebuhr J (২০১১)। "Family Coronaviridae"। King AM, Lefkowitz E, Adams MJ, Carstens EB, International Committee on Taxonomy of Viruses, International Union of Microbiological Societies. Virology Division। Ninth Report of the International Committee on Taxonomy of Viruses। Oxford: Elsevier। পৃষ্ঠা 806–28। আইএসবিএন 978-0-12-384684-6।
- ↑ Sexton NR, Smith EC, Blanc H, Vignuzzi M, Peersen OB, Denison MR (আগস্ট ২০১৬)। "Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens"। Journal of Virology: 7415–28। ডিওআই:10.1128/JVI.00080-16। পিএমআইডি 27279608। পিএমসি 4984655 ।
- ↑ Fehr AR, Perlman S (২০১৫)। "An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization"। Methods in Molecular Biology। Springer: 1–23। আইএসবিএন 978-1-4939-2438-7। ডিওআই:10.1007/978-1-4939-2438-7_1। পিএমআইডি 25720466। পিএমসি 4369385 ।
- ↑ ক খ Fehr AR, Perlman S (২০১৫)। "Coronaviruses: an overview of their replication and pathogenesis"। Methods in Molecular Biology: 1–23। আইএসবিএন 978-1-4939-2437-0। ডিওআই:10.1007/978-1-4939-2438-7_1। পিএমআইডি 25720466। পিএমসি 4369385 ।
- ↑ Snijder EJ, Bredenbeek PJ, Dobbe JC, Thiel V, Ziebuhr J, Poon LL, Guan Y, Rozanov M, Spaan WJ, Gorbalenya AE (আগস্ট ২০০৩)। "Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage"। Journal of Molecular Biology: 991–1004। ডিওআই:10.1016/S0022-2836(03)00865-9। পিএমআইডি 12927536।
- ↑ Simmons G, Zmora P, Gierer S, Heurich A, Pöhlmann S (ডিসেম্বর ২০১৩)। "Proteolytic activation of the SARS-coronavirus spike protein: cutting enzymes at the cutting edge of antiviral research"। Antiviral Research। 100 (3): 605–14। ডিওআই:10.1016/j.antiviral.2013.09.028। পিএমআইডি 24121034। পিএমসি 3889862 ।
See Figure 2.
- ↑ Fehr AR, Perlman S (২০১৫), Maier HJ, Bickerton E, Britton P, সম্পাদকগণ, "Coronaviruses: An Overview of Their Replication and Pathogenesis; Section 4.1 Attachment and Entry", Coronaviruses: Methods and Protocols, Methods in Molecular Biology (ইংরেজি ভাষায়), Springer, 1282, পৃষ্ঠা 1–23, আইএসবিএন 978-1-4939-2438-7, ডিওআই:10.1007/978-1-4939-2438-7_1, পিএমআইডি 25720466, পিএমসি 4369385
- ↑ Fehr AR, Perlman S (২০১৫), Maier HJ, Bickerton E, Britton P, সম্পাদকগণ, "Coronaviruses: An Overview of Their Replication and Pathogenesis; Section 2 Genomic Organization", Coronaviruses: Methods and Protocols, Methods in Molecular Biology (ইংরেজি ভাষায়), Springer, পৃষ্ঠা 1–23, আইএসবিএন 978-1-4939-2438-7, ডিওআই:10.1007/978-1-4939-2438-7_1, পিএমআইডি 25720466, পিএমসি 4369385
- ↑ ক খ গ ঘ Fehr AR, Perlman S (২০১৫)। "Coronaviruses: an overview of their replication and pathogenesis"। Methods in Molecular Biology। 1282: 1–23। আইএসবিএন 978-1-4939-2437-0। ডিওআই:10.1007/978-1-4939-2438-7_1। পিএমআইডি 25720466। পিএমসি 4369385 ।
- ↑ Sexton NR, Smith EC, Blanc H, Vignuzzi M, Peersen OB, Denison MR (আগস্ট ২০১৬)। "Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens"। Journal of Virology। 90 (16): 7415–28। ডিওআই:10.1128/JVI.00080-16। পিএমআইডি 27279608। পিএমসি 4984655 ।
Finally, these results, combined with those from previous work (33, 44), suggest that CoVs encode at least three proteins involved in fidelity (nsp12-RdRp, nsp14-ExoN, and nsp10), supporting the assembly of a multiprotein replicase-fidelity complex, as described previously (38).
- ↑ উদ্ধৃতি ত্রুটি:
<ref>ট্যাগ বৈধ নয়;Fehr_2015নামের সূত্রটির জন্য কোন লেখা প্রদান করা হয়নি - ↑ ক খ Fehr AR, Perlman S (২০১৫)। "Coronaviruses: an overview of their replication and pathogenesis"। Maier HJ, Bickerton E, Britton P। Coronaviruses। Methods in Molecular Biology। 1282। Springer। পৃষ্ঠা 1–23। আইএসবিএন 978-1-4939-2438-7। ডিওআই:10.1007/978-1-4939-2438-7_1। পিএমআইডি 25720466। পিএমসি 4369385 ।
See section: Coronavirus Life Cycle—Assembly and Release
- ↑ "Transmission of Novel Coronavirus (2019-nCoV) | CDC"। www.cdc.gov (ইংরেজি ভাষায়)। ২০২০-০১-৩১। সংগ্রহের তারিখ ২০২০-০২-০১।
- ↑ Masters PS (২০০৬-০১-০১)। "The molecular biology of coronaviruses"। Advances in Virus Research। 66। Academic Press: 193–292। আইএসবিএন 9780120398690। ডিওআই:10.1016/S0065-3527(06)66005-3। পিএমআইডি 16877062।
Nevertheless, the interaction between S protein and receptor remains the principal, if not sole, determinant of coronavirus host species range and tissue tropism.
- ↑ Cui J, Li F, Shi ZL (মার্চ ২০১৯)। "Origin and evolution of pathogenic coronaviruses"। Nature Reviews. Microbiology। 17 (3): 181–192। ডিওআই:10.1038/s41579-018-0118-9। পিএমআইডি 30531947।
Different SARS-CoV strains isolated from several hosts vary in their binding affinities for human ACE2 and consequently in their infectivity of human cells76,78 (Fig. 6b)
- ↑ Li F, Li W, Farzan M, Harrison SC (সেপ্টেম্বর ২০০৫)। "Structure of SARS coronavirus spike receptor-binding domain complexed with receptor"। Science। 309 (5742): 1864–68। ডিওআই:10.1126/science.1116480। পিএমআইডি 16166518। বিবকোড:2005Sci...309.1864L।
- ↑ Wertheim JO, Chu DK, Peiris JS, Kosakovsky Pond SL, Poon LL (জুন ২০১৩)। "A case for the ancient origin of coronaviruses"। Journal of Virology। 87 (12): 7039–45। ডিওআই:10.1128/JVI.03273-12। পিএমআইডি 23596293। পিএমসি 3676139 ।
- ↑ Woo PC, Lau SK, Lam CS, Lau CC, Tsang AK, Lau JH, ও অন্যান্য (এপ্রিল ২০১২)। "Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavirus"। Journal of Virology। 86 (7): 3995–4008। ডিওআই:10.1128/JVI.06540-11। পিএমআইডি 22278237। পিএমসি 3302495 ।
- ↑ Bidokhti MR, Tråvén M, Krishna NK, Munir M, Belák S, Alenius S, Cortey M (সেপ্টেম্বর ২০১৩)। "Evolutionary dynamics of bovine coronaviruses: natural selection pattern of the spike gene implies adaptive evolution of the strains"। The Journal of General Virology। 94 (Pt 9): 2036–49। ডিওআই:10.1099/vir.0.054940-0। পিএমআইডি 23804565।
- ↑ Vijgen L, Keyaerts E, Moës E, Thoelen I, Wollants E, Lemey P, ও অন্যান্য (ফেব্রুয়ারি ২০০৫)। "Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event"। Journal of Virology। 79 (3): 1595–604। ডিওআই:10.1128/jvi.79.3.1595-1604.2005। পিএমআইডি 15650185। পিএমসি 544107 ।
- ↑ Lau SK, Lee P, Tsang AK, Yip CC, Tse H, Lee RA, ও অন্যান্য (নভেম্বর ২০১১)। "Molecular epidemiology of human coronavirus OC43 reveals evolution of different genotypes over time and recent emergence of a novel genotype due to natural recombination"। Journal of Virology। 85 (21): 11325–37। ডিওআই:10.1128/JVI.05512-11। পিএমআইডি 21849456। পিএমসি 3194943 ।
- ↑ Lau SK, Li KS, Tsang AK, Lam CS, Ahmed S, Chen H, ও অন্যান্য (আগস্ট ২০১৩)। "Genetic characterization of Betacoronavirus lineage C viruses in bats reveals marked sequence divergence in the spike protein of pipistrellus bat coronavirus HKU5 in Japanese pipistrelle: implications for the origin of the novel Middle East respiratory syndrome coronavirus"। Journal of Virology। 87 (15): 8638–50। ডিওআই:10.1128/JVI.01055-13। পিএমআইডি 23720729। পিএমসি 3719811 ।
- ↑ Huynh J, Li S, Yount B, Smith A, Sturges L, Olsen JC, ও অন্যান্য (ডিসেম্বর ২০১২)। "Evidence supporting a zoonotic origin of human coronavirus strain NL63"। Journal of Virology। 86 (23): 12816–25। ডিওআই:10.1128/JVI.00906-12। পিএমআইডি 22993147। পিএমসি 3497669 ।
- ↑ Vijaykrishna D, Smith GJ, Zhang JX, Peiris JS, Chen H, Guan Y (এপ্রিল ২০০৭)। "Evolutionary insights into the ecology of coronaviruses"। Journal of Virology। 81 (8): 4012–20। ডিওআই:10.1128/jvi.02605-06। পিএমআইডি 17267506। পিএমসি 1866124 ।
- ↑ Gouilh, Meriadeg Ar; Puechmaille, Sébastien J.; Gonzalez, Jean-Paul; Teeling, Emma; Kittayapong, Pattamaporn; Manuguerra, Jean-Claude (অক্টোবর ২০১১)। "SARS-Coronavirus ancestor's foot-prints in South-East Asian bat colonies and the refuge theory"। Infection, Genetics and Evolution। 11 (7): 1690–1702। ডিওআই:10.1016/j.meegid.2011.06.021। পিএমআইডি 21763784।
- ↑ Cui J, Han N, Streicker D, Li G, Tang X, Shi Z, ও অন্যান্য (অক্টোবর ২০০৭)। "Evolutionary relationships between bat coronaviruses and their hosts"। Emerging Infectious Diseases। 13 (10): 1526–32। ডিওআই:10.3201/eid1310.070448। পিএমআইডি 18258002। পিএমসি 2851503 ।
- ↑ Crossley BM, Mock RE, Callison SA, Hietala SK (ডিসেম্বর ২০১২)। "Identification and characterization of a novel alpaca respiratory coronavirus most closely related to the human coronavirus 229E"। Viruses। 4 (12): 3689–700। ডিওআই:10.3390/v4123689। পিএমআইডি 23235471। পিএমসি 3528286 ।
- ↑ Liu P, Shi L, Zhang W, He J, Liu C, Zhao C, ও অন্যান্য (নভেম্বর ২০১৭)। "Prevalence and genetic diversity analysis of human coronaviruses among cross-border children"। Virology Journal (ইংরেজি ভাষায়)। 14 (1): 230। ডিওআই:10.1186/s12985-017-0896-0। পিএমআইডি 29166910। পিএমসি 5700739 ।
- ↑ ক খ Forgie S, Marrie TJ (ফেব্রুয়ারি ২০০৯)। "Healthcare-associated atypical pneumonia"। Seminars in Respiratory and Critical Care Medicine। 30 (1): 67–85। ডিওআই:10.1055/s-0028-1119811। পিএমআইডি 19199189।
- ↑ Corman VM, Muth D, Niemeyer D, Drosten C (২০১৮)। "Hosts and Sources of Endemic Human Coronaviruses"। Advances in Virus Research। 100: 163–88। আইএসবিএন 978-0-12-815201-0। ডিওআই:10.1016/bs.aivir.2018.01.001। পিএমআইডি 29551135।
বহিঃসংযোগ সম্পাদনা
সরকারি স্বাস্থ্য সংস্থা
- করোনাভাইরাস (কোভিড-১৯) সম্পর্কিত প্রয়োজনীয় তথ্যাবলি, রোগতত্ত্ব, রোগ নিয়ন্ত্রণ ও গবেষণা ইনস্টিটিউট
- বিশ্ব স্বাস্থ্য সংস্থা: কোভিড -১৯ (প্রশ্ন এবং উত্তর) (ইংরেজি) বিশ্ব স্বাস্থ্য সংস্থা কর্তৃক
- কোভিড -১৯(ইংরেজি)(প্রশ্ন এবং উত্তর) (ইংরেজি) ইউরোপীয়ান সেন্টার ফর ডিজিজ প্রিভেনশন অ্যান্ড কন্ট্রোল কর্তৃক
- কোভিড -১৯[স্থায়ীভাবে অকার্যকর সংযোগ] (ইংরেজি) চায়না ন্যাশনাল হেলথ কমিশন কর্তৃক
- কোভিড -১৯ (ইংরেজি) (প্রশ্ন এবং উত্তর) (ইংরেজি) সিঙ্গাপুর মিনিস্ট্রি অব হেলথ কর্তৃক
- কোভিড -১৯ (ইংরেজি)
(প্রশ্ন এবং উত্তর) (ইংরেজি) সেন্টার ফর ডিজিজ কন্ট্রোল অ্যান্ড প্রিভেনশন (যুক্তরাষ্ট্রের সিডিসি)
- করোনাভাইরাস রোগ ২০১৯ (ইংরেজি)
(প্রশ্ন এবং উত্তর) (ইংরেজি) ন্যাশনাল ইন্সটিটিউট ফর অকুপেশনাল সেফটি অ্যান্ড হেলথ কর্তৃক
উপাত্ত ও মানচিত্র
- বিশ্ব স্বাস্থ্য সংস্থা কর্তৃক করোনাভাইরাস রোগ (কোভিড -২০১৯)-এর পরিস্থিতি প্রতিবেদন (দেশ অনুযায়ী নিশ্চিত সংক্রামণের অফিসিয়াল সংখ্যা) (ইংরেজি)
- বিশ্ব স্বাস্থ্য সংস্থা এর দেশ অনুসারে সংক্রামণের মানচিত্র (ইংরেজি)
- করোনাভাইরাস কোভিড-১৯ এর বৈশ্বিক সংক্রামণ (ইংরেজি) এবং ঐতিহাসিক তথ্য (ইংরেজি) জন্স হপকিন্স বিশ্ববিদ্যালয় কর্তৃক
- কোভিড-১৯ এর সাম্প্রতিক সংবাদ ও তথ্যসম্ভার (ইংরেজি) The BMJ কর্তৃক
- কোভিড-১৯ তথ্য কেন্দ্র (ইংরেজি) by The Lancet কর্তৃক
- করোনাভাইরাস তথ্যসম্ভার (ইংরেজি) Cell Press কর্তৃক
- করোনাভাইরাস পর্যবেক্ষণ (ইংরেজি) জন্স হপকিন্স বিশ্ববিদ্যালয় এর তথ্যের ভিত্তিতে
- Google Scholar list of Covid-19 research resources (ইংরেজি), এটি একইসাথে আবার এসব সংস্থার গবেষণাকেন্দ্রগুলোর সাথেও যুক্ত সিডিসি, NEJM, JAMA, The Lancet, Cell, The BMJ, Elsevier, অক্সফোর্ড ইউনিভার্সিটি প্রেস, নেচার পত্রিকা, Wiley, কেমব্রিজ ইউনিভার্সিটি প্রেস, এবং medRxiv
- বৈশ্বিক ভ্রমণ বাঁধানিষেধ (ইংরেজি) ডব্লিউএফপি এর তথ্যের ভিত্তিতে